Alinhamento Múltiplo e Filogenia
O alinhamento múltiplo de sequências tem como principal função estabelecer relações filogenéticas entre diferentes proteínas, através da comparação da proteína de interesse com membros da mesma família de proteínas. Serve também para prever a função de certas estruras secundárias e terciárias de proteínas, definir primers para PCR (Polimerase Chain Reaction), montar (assembly) um genoma, detetar zonas conservadas e entender o funcionamento das taxas de mutação relativa dentro de um grupo de sequências de DNA ou proteína.
Foi obtido o alinhamento de pares e o alinhamento múltiplo da proteína do gene CR1 com nove das sequências homólogas e da proteína do gene CR1 com exclusão da classe dos mamíferos com oito das sequências homólogas previamente encontradas no BLAST, e referidas em ANÁLISE DE HOMOLOGIAS (não se procedeu ao alinhamento de todas as proteínas encontradas devido à sua quantidade ser muito elevada) pelo programa MEGA7.
Para ver o alinhamento múltiplo das proteínas clique no link abaixo:
https://www.dropbox.com/s/3cag4zm8kmienef/finalalinhamentomultiplo.mas?dl=0
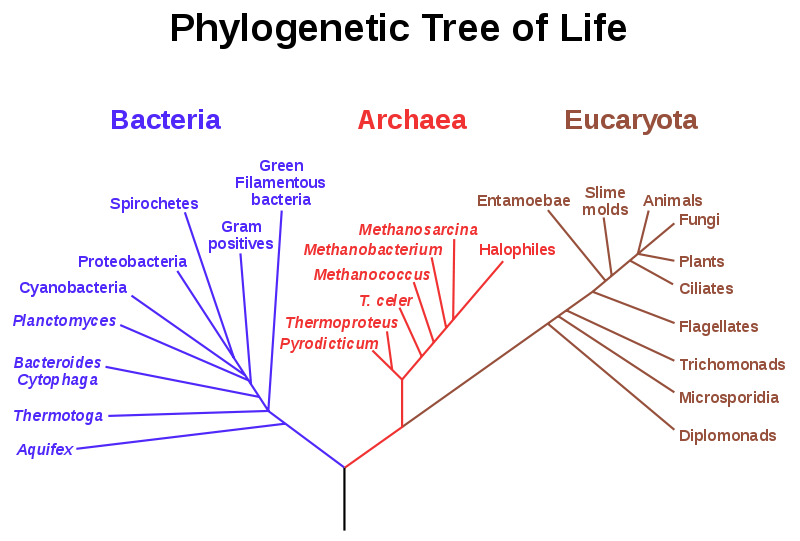
Uma árvore filogenética, também designada por "Árvore da Vida", é uma representação gráfica, em forma árvore, das relações evolutivas entre várias espécies ou outras entidades que possam ter um ancestral comum, sendo que a proximidade na árvore sugere também proximidade evolutiva, em que cada nó indica os ancestrais comuns e o comprimento de cada ramo pode representar estimativas do tempo evolutivo. Cada nó terminal de uma árvore filogenética é chamado de "unidade taxonómica".
A análise filogenética de um conjunto de sequências (DNA, RNA, proteínas) representa o modo de como cada sequência pode ter sido derivada ao longo do processo de evolução, tendo como principais funções:
- Estudar relações evolutivas
que permitam estabelecer relações taxonómicas;
- Determinação da
função de sequências de DNA/ proteínas;
- Identificação de ramos com
motivos específicos que podem ter consequências funcionais para um determinado gene;
- Hierarquização e identificação de mutações numa árvore;
- Identificar efeitos de seleção natural a nível de um gene, podendo esta ser negativa ou positiva;
- Fator determinante em algoritmos de alinhamento múltiplo.
Árvore Filogenética da proteína do gene CR1 (SCR25 - Homo sapiens) obtida pelo Método da Máxima Semelhança, no programa MEGA7:
Em anexo está disponível um ficheiro em Word para download com a legenda e explicação da árvore filogenética obtida e também do método utilizado para a construção da mesma, através do MEGA7:
Árvore Filogenética da proteína do gene CR1 (SCR25 - Homo sapiens) obtida pelo Método do Neighbour-joining, no programa MEGA7:
Em anexo está disponível um ficheiro em Word para download com a legenda e explicação da árvore filogenética obtida e também do método utilizado para a construção da mesma, através do MEGA7:
Árvore Filogenética da proteína do gene CR1 (SCR25 - Homo sapiens) obtida pelo Método do UPGMA, no programa MEGA7:
Em anexo está disponível um ficheiro em Word para download com a legenda e explicação da árvore filogenética obtida e também do método utilizado para a construção da mesma, através do MEGA7:
Árvore Filogenética da proteína do gene CR1 (SCR25 - Homo sapiens) com a exclusão da classe dos mamíferos obtida pelo Método da Máxima Semelhança, no programa MEGA7:
Em anexo está disponível um ficheiro em Word para download com a legenda e explicação da árvore filogenética obtida e também do método utilizado para a construção da mesma, através do MEGA7:
Árvore Filogenética da proteína do gene CR1 (SCR25 - Homo sapiens) com a exclusão da classe dos mamíferos obtida pelo Método do UPGMA, no programa MEGA7:
Em anexo está disponível um ficheiro em Word para download com a legenda e explicação da árvore filogenética obtida e também do método utilizado para a construção da mesma, através do MEGA7:
Os métodos Neighbour Joining e UPGMA (Unweighted Pair Group Method Using Arithmetic Averages) são métodos de distância, ou seja, baseiam‐se na distância entre os diversos pares de sequências considerados, através de uma matriz de distância entre todas as sequências. São métodos bastante fáceis e de rápida realização. Contudo, tem como desvantagens o facto de o resultado nem sempre ser ideal a nível evolutivo, de ser influenciável por taxas de mutação diferentes, assim como os ramos nem sempre representam o conjunto de mutações partilhadas.
Através da análise das diferentes árvores filogenéticas obtidas pelo programa MEGA7, para a proteína do gene CR1, podemos concluir que os resultados obtidos foram bastante semelhantes a nível evolutivo e que não foram encontradas descrepâncias em relação à literatura.
Contudo, para a proteína do gene CR1 com exclusão da classe dos mamíferos os resultados não foram filogenica e evolutivamente concordantes. Isto deve-se ao facto de os scores de homologia serem bastante baixos e algumas das espécies utilizadas para a execução do programa se encontrarem na categoria Predicted .
Referências Bibliográficas:
- Material de apoio à UC de Bioinformática, Aula Teórica 3
