Alterações pós-tradução
As alterações pós-tradução foram estudadas recorrendo a diversas ferramentas, sendo que a primeira abordada foi o PDB. Foi possível observar a partir deste site modificações moleculares assim como as modificações dos aminoácidos. Na imagem que se segue verifica-se que a proteína apresenta um peptídeo de sínal com 42 a.a. e que a restante cadeia polipeptidica é sujeita a modificações.
O documento acima, passível de ser descarregado, permite observar as acima referidas modificações dos aminoácidos. Observa-se que estas se tratam pontes disulfureto e glicosilações em a.a.
- Glicosilação:
Processo específico, que occore apenas no retículo endoplasmático e complexo de golgi, de adição de oligosacarídeos a cadeias proteicas. Proteínas citoplasmáticas não estão, em princípio, sujeitas a esta reação. É uma modificação primordial para a formação de proteínas de membrana e secretórias e para o desempenho de funções como reconhecimento celular, adesão, entre outras, pela proteína.
É possível observar gráficamente este processos pelo NetNGlyc 1.0 Server:

- Fosforilação
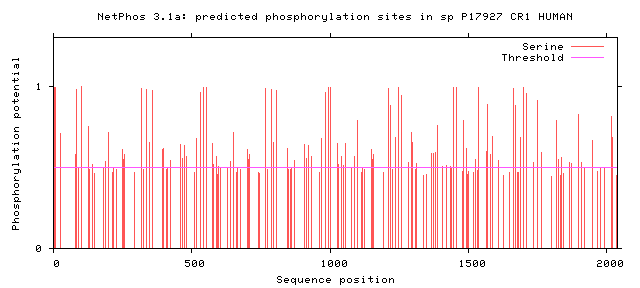
Processo de adição de um grupo fosfato (PO4) terminal do ATP para um grupo hidroxila da cadeia lateral de uma serina, treonina ou tirosina, sendo a reação catalisada por uma proteína-cinase. É um método comum de regulação de uma proteína em que a adição ou remoção do grupo fosfato leva a ativação ou inibição da mesma.
Devido ao elevado número de a.a. fosforilados decidiu-se separar os resultados nos três gráficos que se seguem, referentes à serina, treonina e tirosina, respetivamente.


- Hidroxilação:
- Acetilação:
Processo que permite a adição de um grupo acetilo (CH3CO) em proteínas. A presença destes grupos pode levar a ativação ou inativação de proteínas, para além de que a acetilação do terminal amino de uma proteína pode funcionar como sinal de degradação. Este processo pode ainda regular fatores de transcrição, proteínas efetoras, e proteínas do citoesqueleto.
Foi utilizada a ferramenta NetAcet 1.0 Server para prever se esta modificação ocorria na proteína em questão. Como o score é inferir a 0,5, consideramos que a proteína não apresenta acetilação.
- Metilação:
Processo de transferência de um grupo metilo (CH3) de um carbono ou oxigénio. Ocorre apenas em lisinas e argininas e leva a um aumento da hidrofobicidade da proteína, que pode neutralizar a carga negativa de aminoácidos, quando ligados a ácidos carboxílicos.
Pela ferramenta PMes, é possivel prever que não ocorrem metilações em lisinas, apenas em argininas. Selecionando um limite inferior de probabilidade de ocorrer metilação de 0,6, para evitar falsos positivos, obtém-se a tabela seguinte:
Referências Bibiiográficas:
- Material de apoio à UC de Bioinformática, Aula Teórica 5
- Wikipedia
- Livro Molecular Biology Of The Cell 5º edição
- https://www.ifsc.usp.br/~rdemarco/FFI0760/modificacaodeproteinas.pdf
